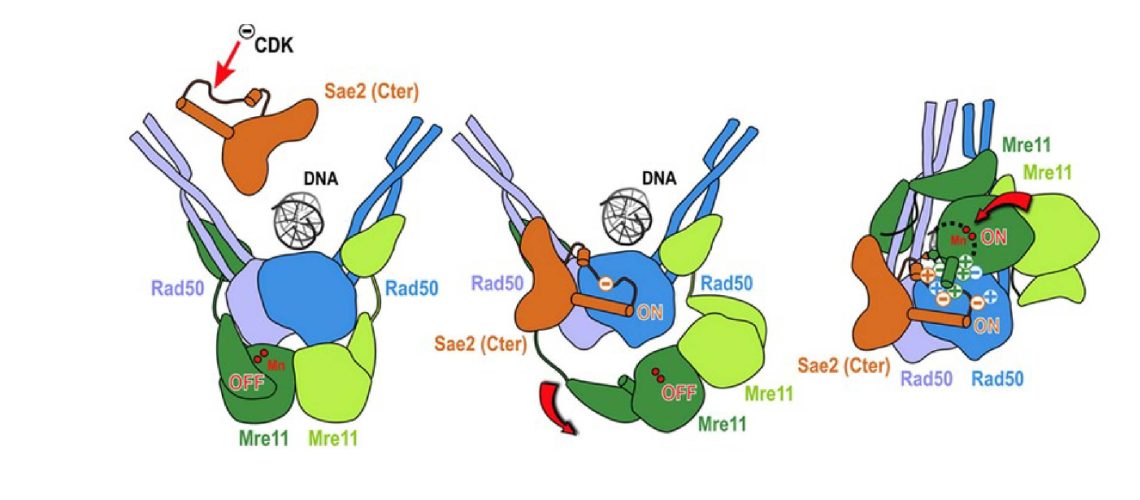
Un nuovo studio pubblicato dal laboratorio Cejka e collaboratori su “Molecular Cell” fornisce informazioni sui meccanismi molecolari responsabili dell’attività endonucleasica di Mre11-Rad50 da parte di Sae2/CtIP.
Bellinzona, 13 giugno 2024 – In Saccharomyces cerevisiae (S. cerevisiae), l’attività nucleasica di Mre11-Rad50-Xrs2 (MRX)-Sae2 è necessaria per la resezione delle rotture del DNA con strutture secondarie o blocchi proteici, mentre nell’uomo, l’omologo MRE11-RAD50-NBS1 (MRN) con CtIP è necessario per avviare la resezione delle estremità del DNA in seguito a tutti i tipi di rotture. Sae2/CtIP fosforilato stimola l’attività endonucleasica di MRX/N. Le informazioni strutturali sull’attivazione della nucleasi Mre11 sono disponibili solo per organismi privi di Sae2/CtIP, quindi si sa poco su come Sae2/CtIP attivi l’”ensemble” delle nucleasi. Utilizzando una combinazione di modellazione strutturale con AlphaFold2 e di saggi biochimici e genetici, gli autori svelano il meccanismo di attivazione di Mre11 da parte di Sae2. Mostrano come Sae2 fosforilato stabilizzi la nucleasi Mre11 nel suo stato attivo. In aggiunta, l’utilizzo di mutazioni compensatorie rivelano come Sae2 attivi MRX in vitro e in vivo, supportando il modello strutturale. Infine, lo studio rivela come CtIP umano, nonostante la notevole divergenza di sequenza, utilizzi un meccanismo simile per attivare MRN e mette in evidenza i riarrangiamenti strutturali che potrebbero essersi verificati durante l’evoluzione da un antenato comune di Sae2 e CtIP.
Elda Cannavo, del laboratorio Cejka (Istituto per la Ricerca in Biomedicina di Bellinzona, affiliato con la Facoltà di Scienze Biochimiche dell’Università della Svizzera italiana) è uno dei tre autori principali dello studio e Petr Cejka è il coordinatore scientifico insieme a Valérie Borde (Institut Curie, Parigi, Francia) e Raphaël Guerois (Institute for Integrative Biology of the Cell, Parigi, Francia).